共计 1604 个字符,预计需要花费 5 分钟才能阅读完成。
µFormer 是一种用于突变效应预测的两步解决方案,即预测突变蛋白质序列的适应度得分。µFormer 由一个自监督蛋白质语言模型和一组监督评分模块组成。
首先,通过在大量未标记蛋白质序列数据集上预训练掩蔽蛋白质语言模型 (PLM)。在预训练阶段,蛋白质语言模型使用从 UniRef50 收集的超过 3000 万个蛋白质序列进行训练。通过采用掩蔽语言建模策略,蛋白质语言模型学会在给定蛋白质序列中其余残基的情况下预测目标位置上最可能的氨基酸。
其次,通过使用集成到预训练模型中的三个评分模块预测适应度得分。这些模块(残差级、基序级和序列级)捕获蛋白质序列的不同方面,并结合它们的输出以生成最终的适应度得分。该模型使用已知的适应度数据进行训练,最大限度地减少预测分数和实际分数之间的误差。
此外,µFormer 与强化学习 (RL) 策略相结合,可有效探索可能突变的广阔空间。该框架中的蛋白质工程问题被建模为马尔可夫决策过程 (MDP),并使用近端策略优化 (PPO) 来优化突变策略。在突变搜索过程中添加狄利克雷(Dirichlet)噪声,保证有效搜索,避免局部最优。
在不同任务上的表现优于同类方法
研究证明,µFormer 可以处理各种具有挑战性的场景,包括有限数量的测量、同源物很少的孤蛋白、具有多点突变的复杂变体、插入和删除,以及表现出训练数据中不存在的过度激活的突变体。大量实验表明,µFormer 在不同任务上的表现优于同类方法。
为了评估 µFormer 在适应度景观建模和突变效应预测方面的能力,研究人员将其与十种替代方法进行了对比,包括基于 MSA 的方法、基于语言模型的零样本方法和基于学习的方法。
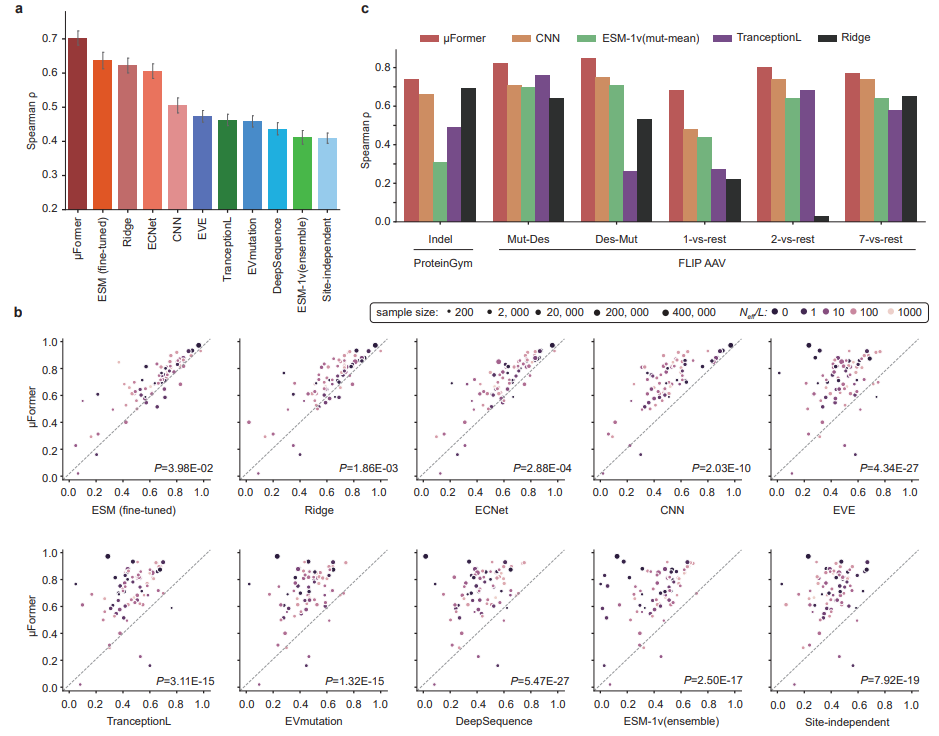
图示:µFormer 与最先进的突变效应预测方法的定量比较。(来源:论文)
首先在 ProteinGym 上评估了所有模型。在所有模型中,µFormer 在不同数据集中预测突变效应的能力最强。平均 Spearman 相关性为 0.703,在 78 个测试数据集中的 48 个上取得了最佳性能。
进一步的分析表明,µFormer 对训练数据大小和同源序列号的变化不敏感,表明 µFormer 是蛋白质突变效应预测的通用工具。
接下来,评估了 µFormer 在插入和删除 (indel) 预测方面的表现。研究人员对 µFormer 在 indel 任务上的表现与四种替代方法进行了基准测试。µFormer 在两个具有 indel 突变的基准数据集上始终优于其他方法。
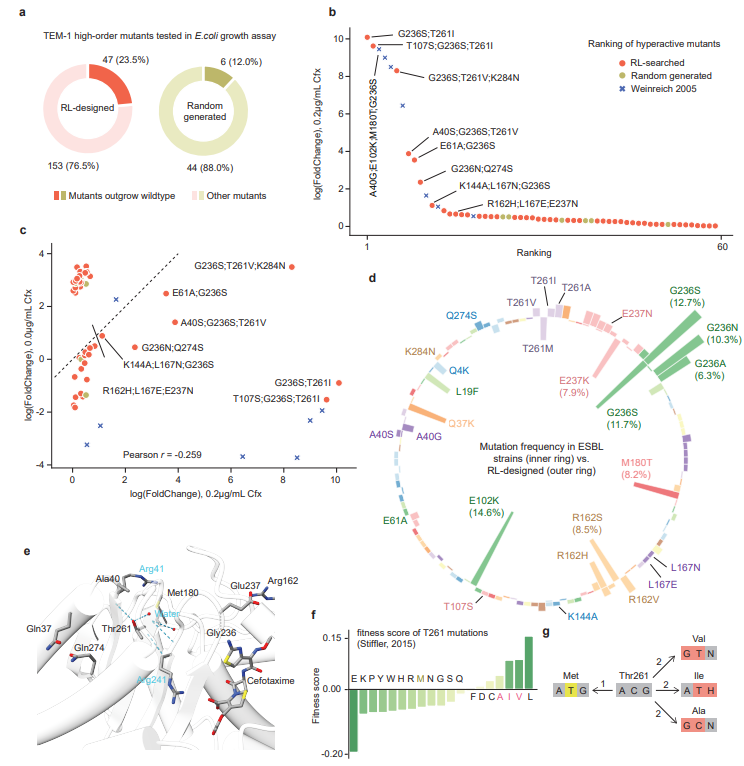
图示:使用 µFormer 和强化学习设计高功能序。(来源:论文)
更重要的是,观察到在使用单个突变体进行训练时,µFormer 在高阶突变效应预测方面表现出色,将 µFormer 部署为「导航」蛋白质设计的通用工具,并结合强化学习,可以在广阔的适应度景观中进行有效的序列搜索。
通过使用此流程对涵盖数万亿个突变候选物的突变空间进行了有效而全面的探索,研究人员设计了一种 β-内酰胺酶来水解新的底物分子,并确定了许多具有多达 3 个点突变的变体,这些变体可能对新底物的活性表现出显著增强。
具体来说,通过仅基于单点突变数据训练模型并探索多点高适应度突变,研究人员在湿实验室实验中测试的 200 个样本中发现了 47 个活性高于野生型的突变。其中包括酶活性比之前发现的最高突变(1000 倍)高 2000 倍的 β-内酰胺酶变体,可作为抵抗耐药性的预防措施。
µFormer 利用预训练的大型蛋白质序列模型,在多个不同的预测任务中取得了出色的表现。可以预见,深度学习模型将进一步加速蛋白质适应度的研究,并为生物药物设计、蛋白质疫苗优化和蛋白质工程等各个领域做出贡献。
参考内容:%C2%B5former-a-deep-learning-framework-for-efficient-protein-fitness-prediction-and-optimization/





