共计 963 个字符,预计需要花费 3 分钟才能阅读完成。
蛋白质在生物体内扮演着不可或缺的角色,准确预测其功能对于实际应用至关重要。尽管高通量技术促进了蛋白质序列数据的激增,但揭示蛋白质的确切功能仍然需要大量时间和资源。目前,许多方法都依赖于蛋白质序列进行预测,而针对蛋白质结构的方法很少。
为了应对这些挑战,东北大学的研究人员从蛋白质结构出发,提出将卷积神经网络 (CNN)和图卷积网络 (GCN)结合成一个统一框架,称为双模型自适应权重融合网络 (Two-model Adaptive Weight Fusion Network,TAWFN),用于蛋白质功能预测。
TAWFN 在预测蛋白质结构功能方面表现出了良好的性能,优于现有方法。
相关研究以「TAWFN: a deep learning framework for protein function prediction」为题,于 9 月 23 日发布在《Bioinformatics》上。

论文链接:
单独使用 CNN、GCN 预测蛋白功能的缺陷
准确识别蛋白质功能有助于更深入地了解疾病机制,并有望发现新的治疗靶点。
开发一种准确有效的蛋白质功能预测方法至关重要。当前预测蛋白质功能的方法主要集中在三个领域:蛋白质序列、蛋白质结构和蛋白质-蛋白质相互作用网络。
当前针对蛋白质结构的方法很少,通常单独使用卷积神经网络(CNN) 或图卷积网络 (GCN)。单独使用 CNN 或 GCN 存在以下问题:
TAWFN:用于蛋白质功能预测
为了解决这些问题,东北大学研究人员提出了一种新型蛋白质功能预测方法TAWFN。该方法集成了 CNN 和 GCN,同时利用了蛋白质结构和蛋白质语言模型。对蛋白质结构进行处理,得到相应的蛋白质序列。
研究的主要贡献总结如下:
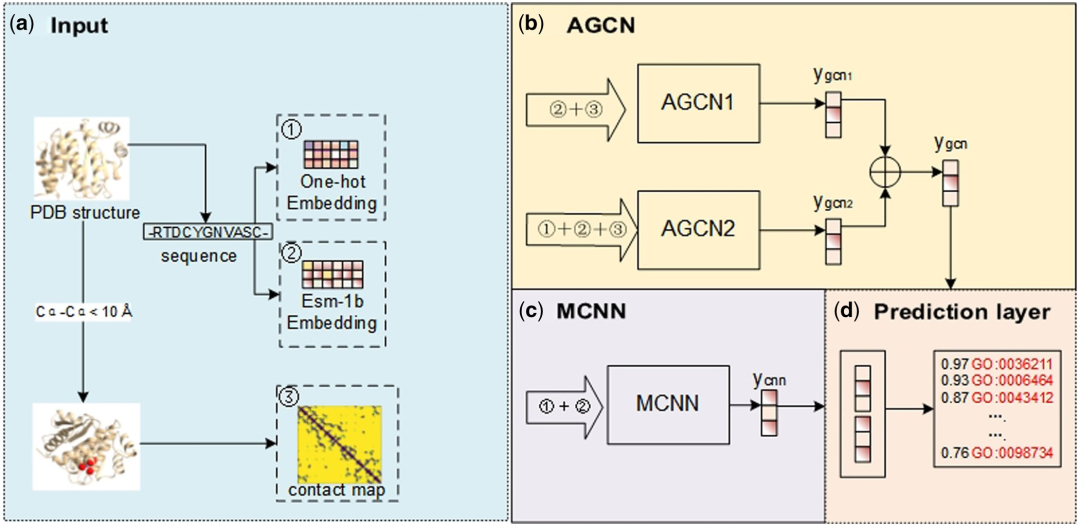
具体而言,TAWFN 的结构如下图所示,主要由四个模块组成:(1)输入数据生成模块:该模块生成蛋白质接触图和序列编码特征,包括 ESM-1b 编码和独热编码。(2)基于 GCN 的 AGCN 模块:该模块包括两个子模块 AGCN1和 AGCN2,两个子模块在处理不同的输入时共享同一个 AGCN 网络。它产生初步的预测结果
。(3)基于 CNN 的 MCNN 模块:该模块生成初步的预测结果
。(4)自适应融合模块:该模块通过计算将两个初步预测结果,
和
融合,生成最终的预测分数。