共计 1212 个字符,预计需要花费 4 分钟才能阅读完成。
基于扩散的生成模型近期在从头设计蛋白质方向取得重大突破,显著推动了领域发展。
然而,有证据表明:一些特定折叠类别的蛋白质骨架,AI 模型仍难以生成。并且,当前广泛使用的可设计性、新颖性和多样性等指标,均无法反映生成结果对天然蛋白质空间的覆盖程度。
为此,近期来自清华大学、北京生命科学研究所等机构的研究团队提出了一种无监督系统 ——TopoDiff 框架,通过学习和利用全局几何感知的潜在表征,实现了基于扩散模型的无条件与可控蛋白质生成。研究论文发表于《Nature Machine Intelligence》。

论文地址:https://www.nature.com/articles/s42256-025-01059-x
提升蛋白质折叠模式覆盖度
为了提升生成样本对特定蛋白质折叠的覆盖率,有方法采用残基级别的一维 / 二维折叠条件约束配合额外微调来生成具有可变环区的免疫球蛋白结构域,或用特定蛋白质类别训练分类器。这些方法虽能增强特定蛋白质类群的覆盖率,但其适用前提是目标类群必须具有清晰明确的定义且包含足量训练样本。
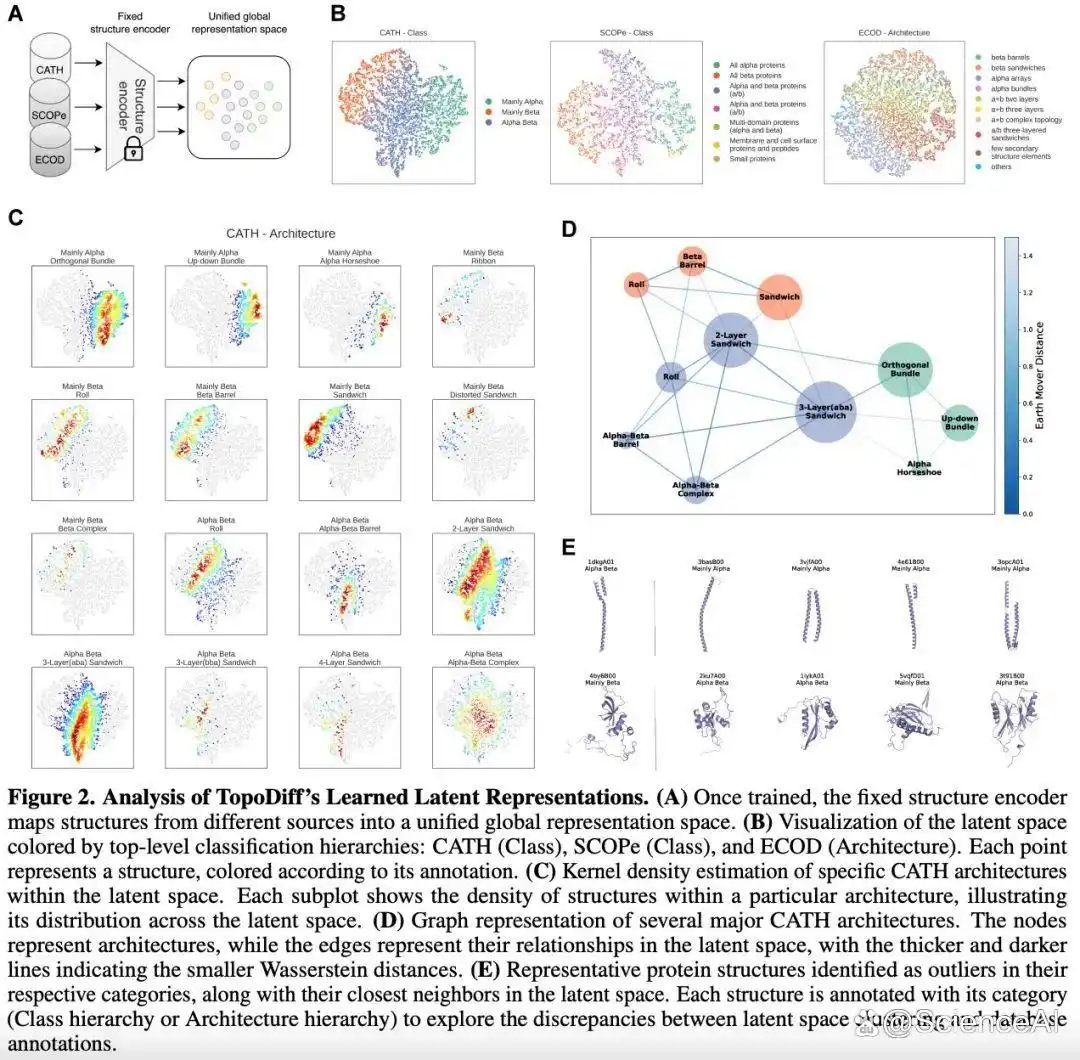
因此,研究团队聚焦于一个重要且通用的无监督学习问题:给定任意数据集,如何在不依赖标注信息或先验认知的条件下训练扩散模型以捕捉潜在数据分布?更进一步,能否利用 SOTA 生成模型的设计优势来学习可解释的潜在编码,从而深化理解?
基于上述问题,研究团队提出 TopoDiff 框架,同步训练基于扩散的结构生成模型与编解码器架构的结构编码器,旨在实现双重目标:
学习能编码蛋白质高级全局几何特征的紧凑、定长连续潜在空间;
构建一个可在残基层面运行的生成模块(以给定的潜在信息为条件进行生成)。
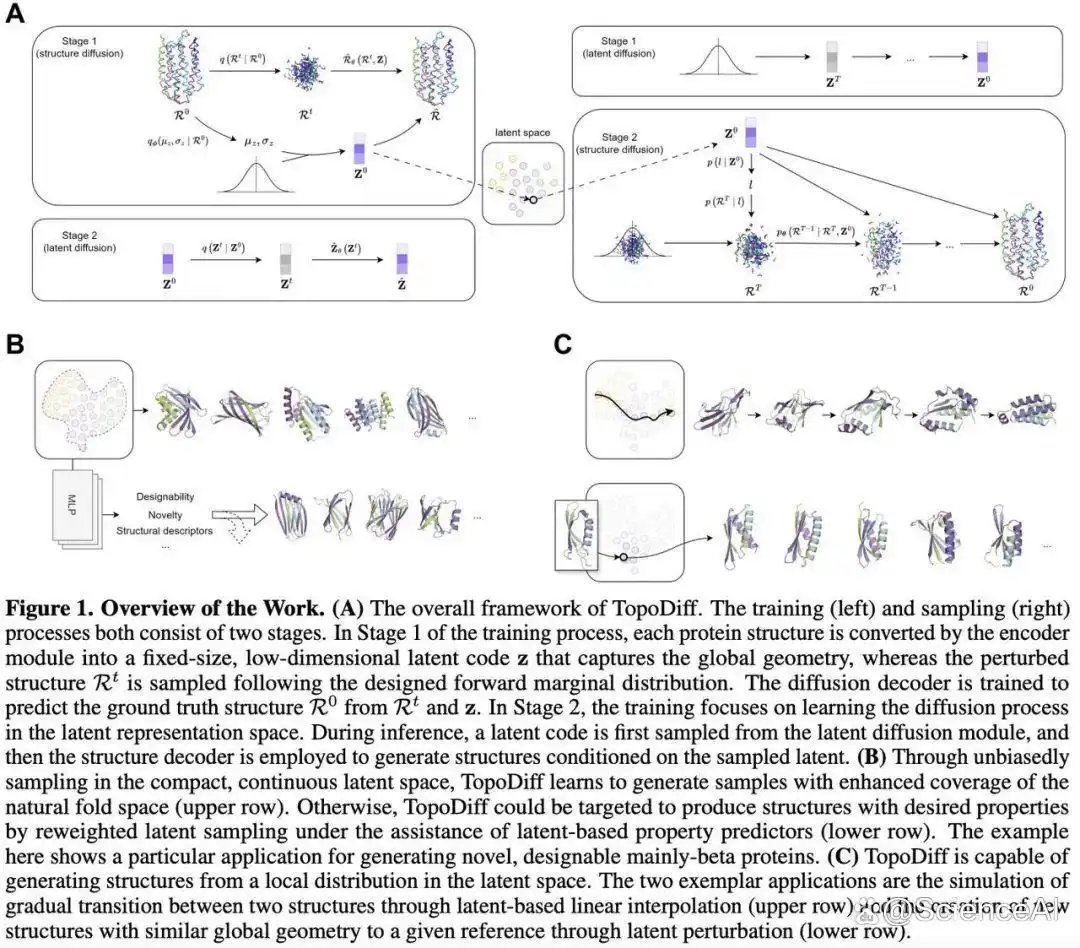
该研究训练了一个简单的潜在扩散模型来无偏地采样学得的潜在分布,并利用采样得到的潜在变量指导蛋白质结构采样 —— 该方案有效提升了数据集中折叠模式的覆盖广度,也为可控生成开辟了新维度。
实验评估
为了评估 TopoDiff 无条件生成新蛋白质结构的性能表现,该研究将 TopoDiff 与多种基于扩散的前沿生成模型进行对比,包括 Genie、FrameDiff、Chroma 和 RFDiffusion。
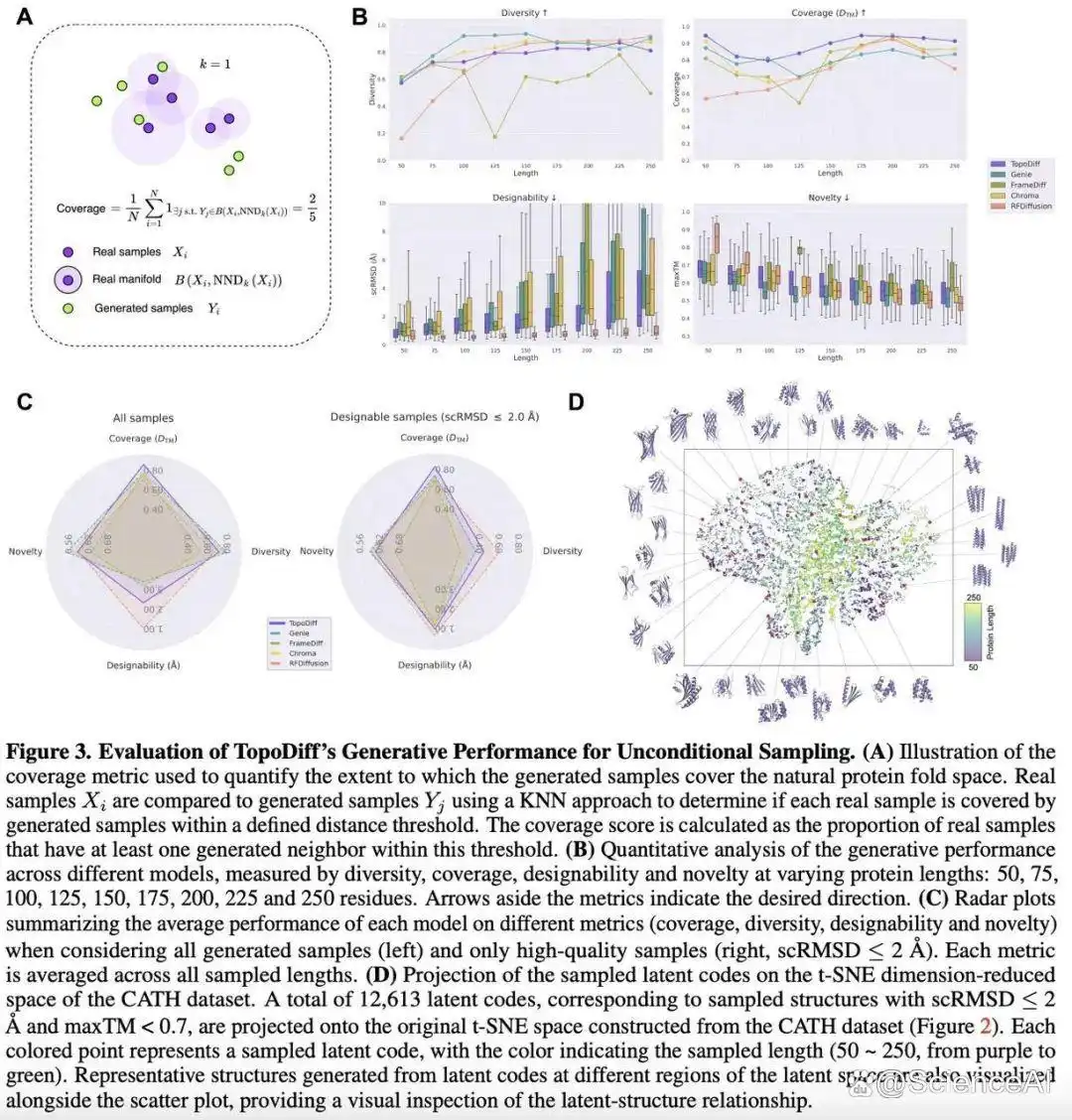
研究团队注意到,现有评估指标主要关注单样本质量(如可设计性、新颖性)或样本内多样性,缺乏对生成样本覆盖已知折叠空间程度的量化。
覆盖度能客观反映模型在现有数据中进行无偏采样的能力。有研究证据表明:忽视这一指标会导致模型选择偏差。
事实上,过去十年中,蛋白质从头设计领域主要局限于 α 螺旋束和 α-β 夹心结构,并且基于扩散的模型尚未能有效扭转这一局面。
因此,研究团队定义了覆盖度指标,并采用该指标及可设计性、新颖性、多样性等传统指标对 TopoDiff 及其他前沿模型进行了系统评估。
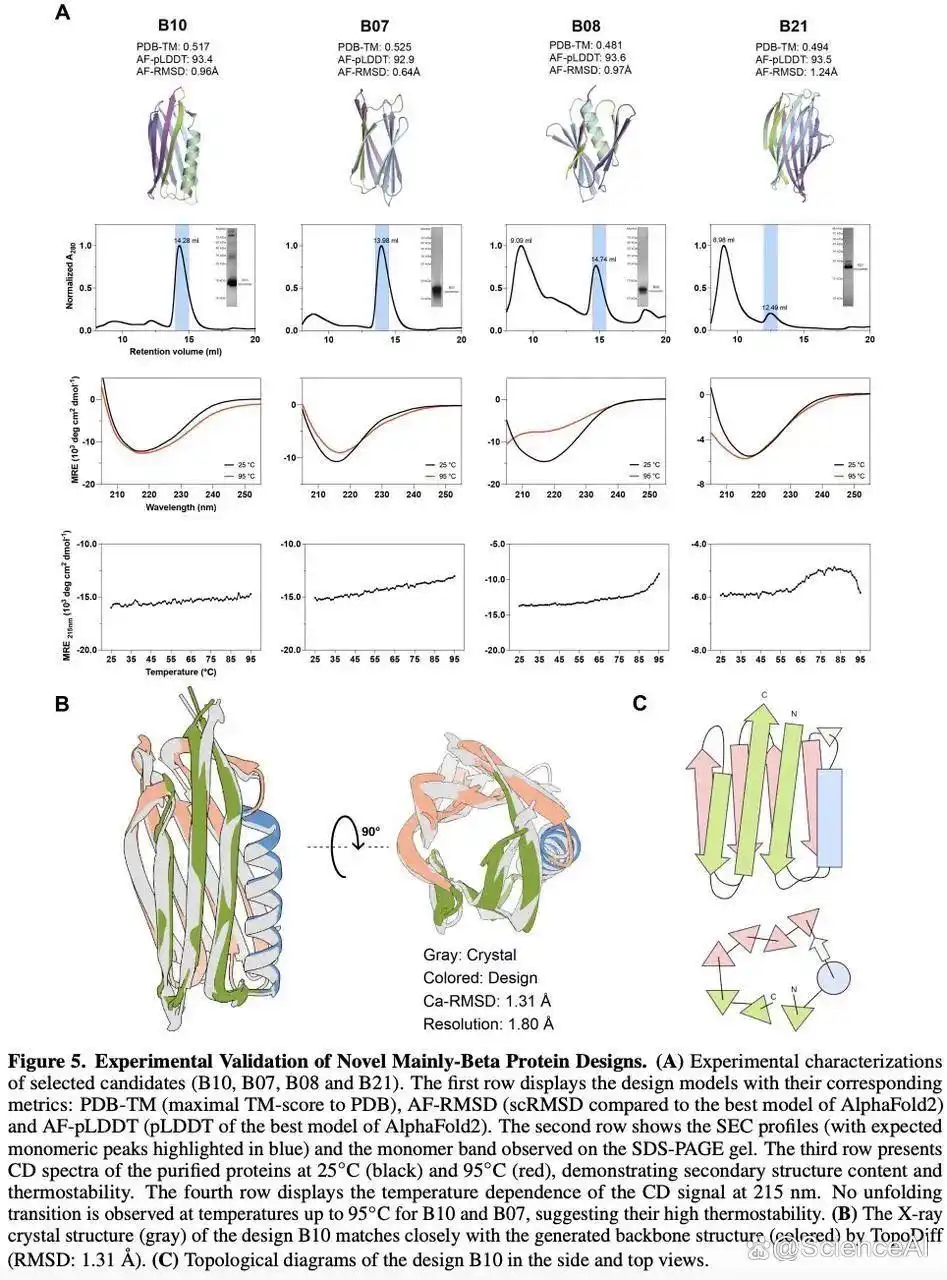
最终,该研究通过生物实验证实:TopoDiff 有效提升了对天然蛋白质折叠模式的覆盖范围。