共计 1489 个字符,预计需要花费 4 分钟才能阅读完成。
编辑 | 萝卜皮
翻译延伸对于维持细胞蛋白质稳态至关重要,并且翻译景观的改变与一系列疾病相关。核糖体分析可以在基因组规模上详细测量翻译。然而,目前尚不清楚如何从这些数据中的技术产物中分离出生物变异,并识别翻译失调的序列决定因素。
在最新的研究中,中国科学院、哈佛大学(Harvard University)、斯坦福大学(Stanford University)、约翰霍普金斯大学(Johns Hopkins University)的研究团队开发了 Riboformer,一个基于深度学习的框架,用于对翻译动态中上下文相关的变化进行建模。
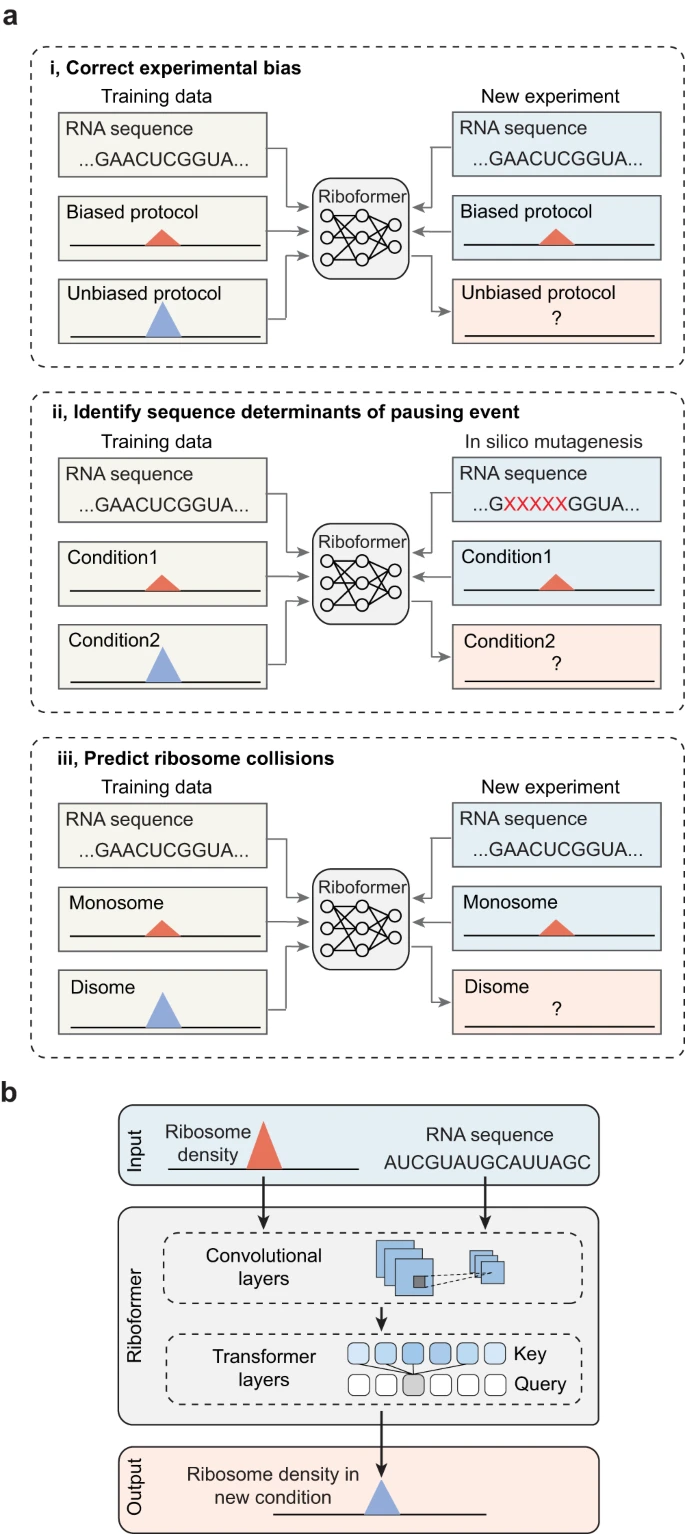
Riboformer 利用 Transformer 架构,能够以密码子分辨率准确预测核糖体密度。当在无偏数据集上进行训练时,Riboformer 会纠正以前未见过的数据集中的实验伪影,这揭示了同义密码子翻译中的细微差异,并揭示了翻译延伸的瓶颈。
研究人员表明 Riboformer 可以与计算机诱变相结合,以识别有助于核糖体在各种生物环境(包括衰老和病毒感染)中停滞的序列基序。
该研究以「Riboformer: a deep learning framework for predicting context-dependent translation dynamics」为题,于 2024 年 3 月 5 日发布在《Nature Communications》。

翻译动态分析的挑战性
核糖体以不同的速率沿着 mRNA 移动,这会影响蛋白质稳态和细胞功能。转录组的延伸率是由局部序列特征(例如 mRNA 二级结构、带电氨基酸簇和连续脯氨酸残基)与全局因素(例如细胞资源可用性和蛋白质质量控制)之间复杂的相互作用决定的。
这些错综复杂的问题会影响翻译效率、共翻译蛋白质折叠和共价修饰。细胞必须微调延伸率,以达到每个 mRNA 的适当蛋白质输出水平,满足调节和蛋白质折叠的需求。但是在理解翻译动力学方面,破译复杂疾病中翻译失调和蛋白质稳态崩溃的调控密码仍然具有挑战性。
核糖体分析的出现使科学家对 mRNA 翻译的理解取得了实质性进展。核糖体分析捕获并测序受核糖体保护免于核酸酶消化的 mRNA 片段,从而可以可靠地推断每个足迹中的核糖体解码位点,并产生有关每个基因的 mRNA 上核糖体分布的信息。
一般来说,密码子上的核糖体密度越大,解码速度就越慢。科学家发现非最佳密码子具有更高的核糖体密度,且解码速度更慢。科学家已经开发了几种计算方法,但人们很少致力于对翻译动态中上下文相关的变化进行建模。并且,现有的计算工具缺乏使用多个数据集(有偏与无偏)来模拟这些伪影引起的核糖体分布变化的能力。
其次,在复杂的生理状态下驱动翻译景观变化的潜在机制,在很大程度上仍然难以捉摸。即便以疾病为中心的研究通常采用病例与对照等设计原则,但目前的方法并未利用这些方法来揭示影响疾病进展中翻译延伸的序列特征。另外,当前模型的预测能力有限。训练后的模型不能用于改进现有实验的分析或预测新环境中的核糖体分布。
深度学习框架 Riboformer
为了应对这些挑战,中国科学院、哈佛大学、斯坦福大学、约翰霍普金斯大学的研究人员提出了 Riboformer,这是一种基于深度学习的框架,可以模拟密码子分辨率下核糖体动力学中上下文相关的变化。
该模型比较了两个数据集之间的核糖体分布,并提取驱动它们之间差异的序列特征。这种结构使经过训练的 Riboformer 模型能够从输入数据集中消除实验偏差,查询核糖体密度相对变化的序列决定因素,并根据单体谱预测核糖体碰撞(二体)位点。