共计 2137 个字符,预计需要花费 6 分钟才能阅读完成。
逆蛋白质折叠(IPF)可以生成有效的氨基酸序列,使其能够折叠成所需的三维骨架结构,从而构建具有特定功能的新蛋白质,可以用于治疗性蛋白质工程、先导化合物优化和抗体设计。
虽然在人工智能的加持下该领域发展迅速,但是挑战依然存在,例如难以预测结构不确定性较高的元素(包括无序区域)。
为了解决此类低置信度残基预测问题,谢菲尔德大学(University of Sheffield)、 阿斯利康(AstraZeneca)的研究人员提出了一个基于掩模先验的去噪扩散 (MapDiff) 框架,该框架能够准确捕捉蛋白质逆折叠的结构信息和残基相互作用。
MapDiff 是一个离散扩散概率模型,它以给定的蛋白质骨架为条件,迭代生成噪声较低的氨基酸序列。在四个具有挑战性的序列设计基准测试中的评估表明,MapDiff 的表现显著优于最先进的方法。此外,MapDiff 生成的计算机模拟序列与不同蛋白质家族和架构中天然蛋白质的物理化学和结构特征非常相似。
该研究以「Mask-prior-guided denoising diffusion improves inverse protein folding」为题,于 2025 年 6 月 16 日发布在《Nature Machine Intelligence》。

蛋白质是由线性氨基酸(AA)序列折叠而成的复杂三维(3D)结构。它们在几乎所有生物过程中都发挥着重要作用,包括新陈代谢、免疫反应和细胞周期调控。
逆蛋白质折叠(IPF)问题是计算生物学和医学中一个基于结构的基本蛋白质设计问题。但是当前仍然面临:传统物理学方法计算成本高且准确性有限,现有机器学习方法对高结构不确定性的残基等测不准的问题。
MapDiff 架构
谢菲尔德大学的研究人员提出了一个基于掩码先验引导的去噪扩散(Mask-prior-guided denoising diffusion,MapDiff)框架,用于准确捕捉结构到序列的映射关系,从而进行 IPF 预测。

图示:用于逆蛋白质折叠的 MapDiff。(来源:论文)
与以往基于图的方法不同,MapDiff 框架将 IPF 预测公式化为去噪扩散问题。扩散过程根据转移概率矩阵逐步向原始 AA 序列添加随机离散噪声,以促进去噪网络的训练。
在去噪过程中,该去噪网络基于三维结构信息,对含噪、随机采样的 AA 序列进行迭代去噪,从而预测或重建原生AA序列。扩散和去噪过程交替迭代,从原生序列的复杂分布中捕捉其采样多样性,并细化预测的 AA 序列。
研究人员提出了一种掩模先验引导的去噪网络,通过每个迭代去噪步骤中的三个操作自适应地调整离散去噪轨迹以生成更有效的 AA 序列。
第一步,基于结构的序列预测器采用等变图神经网络 (EGNN) 对以主干结构为条件的噪声序列进行去噪。
第二步,研究人员使用基于熵的掩码策略和掩码比率适配器,在第一步操作中识别并掩码去噪序列中置信度较低或不确定(例如,结构未确定)的残基,从而生成掩码序列。
第三步,预训练的掩蔽序列设计器网络预测掩蔽残基,以获得其精确的预测结果。掩蔽序列设计器的预训练在扩散和去噪过程之前进行,借助于使用掩蔽语言模型的不变点注意 (IPA) 网络,结合先验结构和序列知识。
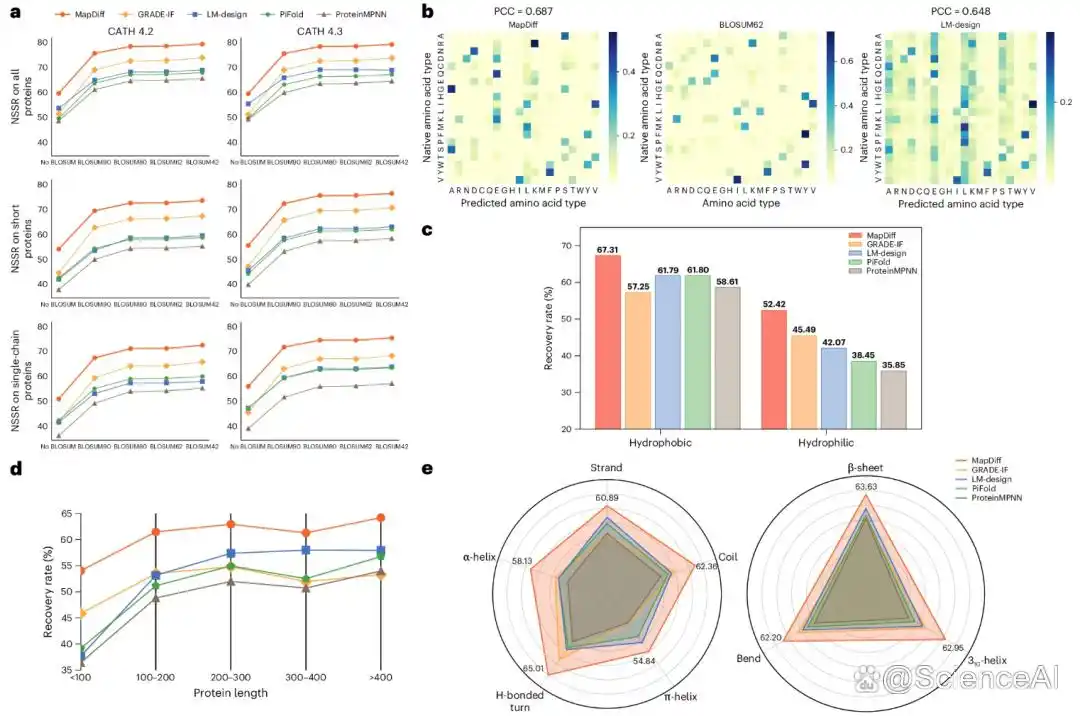
图示:在 CATH 数据集上不同场景下的模型性能比较和敏感性分析。(来源:论文)
基于结构的序列预测器和掩蔽序列设计器利用结构信息和残基相互作用来优化去噪轨迹,从而降低了低置信度残基预测的预测误差。为了高效地生成序列,去噪网络使用非自回归解码以一次性方式生成序列。
为了进一步提升去噪速度和不确定性估计,研究人员在离散生成过程中将 DDIM 与蒙特卡洛 dropout 相结合。DDIM 通过跳过多个去噪步骤来加速序列生成,而蒙特卡洛 dropout 则通过在推理过程中启用 dropout 执行多个随机前向传递来降低不确定性。
性能评估
研究人员将 MapDiff 与最先进的 IPF 预测方法进行了性能比较。结果显示,MapDiff 在多个基准测试和场景中始终优于其他 IPF 模型,甚至优于那些结合外部知识的方法。同时,生成的蛋白质序列与其天然对应序列表现出高度的相似性。
当研究人员使用 AlphaFold2 将 MapDiff 生成的序列折叠回 3D 结构时,即使在序列恢复率较低的情况下,这种 AlphaFold2 折叠结构也与天然蛋白质模板高度相似。

图示:比较 PDB ID 为 1NI8、2HKY 和 2P0X 的蛋白质的三种重新折叠结构(左)和相应的模型设计序列(右)。(来源:论文)
研究人员还进行了一项全面的消融研究,用于分析不同模型组件对预测结果的重要性。即使在训练数据有限的情况下,MapDiff 也表现出生成新蛋白质序列的可转移性和稳健性。
未来方向
研究人员在论文里表示,未来他们会验证 MapDiff 在从头抗体设计和蛋白质工程等实际领域的适用性:将结构预测模型的预测结构作为增量训练的外部数据,整合物理信息约束,利用蛋白质语言模型的顺序进化知识进一步细化残基预测,并通过进行折叠模拟或分子动力学模拟进一步验证设计序列的可折叠性。
论文链接:https://www.nature.com/articles/s42256-025-01042-6
