共计 3236 个字符,预计需要花费 9 分钟才能阅读完成。
基因组可以比喻为细胞「指令操作指南」,这本由 DNA 写就的「天书」详细记载了生物体的全套组装说明书。从外貌特征到内部功能,从生长发育到传宗接代,它事无巨细地掌控着生命活动的每个环节。
有趣的是,这本「天书」里哪怕出现个把字母的排版错误(我们往往称之为变异),都可能让生物体对环境的态度「判若两人」,会瞬间丧失或者产生对某些疾病的抵抗力,变异带来的可能是新加的「技能点」也可能是糟糕的「Debuff」。
可以看出,这本「天书」异常复杂,解码基因组指令在分子水平上是如何被读取,一直是生物学界的最大谜团之一。
根据 DNA 序列预测功能基因组测量值的深度学习模型是破译遗传调控密码的有力工具。现有方法在输入序列长度和预测分辨率之间权衡取舍,从而限制了其模态范围和性能。
在最新的研究中,谷歌 DeepMind 的研究人员提出了 AlphaGenome,它以 1 兆碱基的 DNA 序列作为输入,能够预测数千个功能基因组轨迹,最高可达单碱基对分辨率,涵盖多种模态,包括基因表达、转录起始、染色质可及性、组蛋白修饰、转录因子结合、染色质接触图谱、剪接位点使用情况以及剪接连接点的坐标和强度。
「这将是一个极其有效的工具!」纪念斯隆凯特琳癌症中心的系统生物学家 Caleb Lareau 说道,他率先尝试了这项技术,「这是对人类基因组中 30 亿个字母序列的所有可能变化进行注释和解释的最全面的尝试。这是我们迄今为止拥有的最强大的计算机模拟工具。」
相关研究的预印版以「AlphaGenome: advancing regulatory variant effect prediction with a unified DNA sequence model」为题,于 2025 年 6 月 25 日发布。

论文链接:https://storage.googleapis.com/deepmind-media/papers/alphagenome.pdf
背景
解读基因组序列变异的影响仍然是生物学领域的一大核心挑战。非编码变异位于蛋白质编码区之外,由于其可能引发多种维度的分子结果,因此解读起来尤其困难。
例如,非编码变异可以调节基因组特性,例如染色质的可及性、表观遗传修饰和染色质的三维构象。变异可以通过改变表达水平或通过剪接改变序列组成来进一步影响信使 RNA(mRNA)的可用性。此外,变异还可能表现出细胞类型或组织特异性效应。
目前,科学家所观察到的人类遗传变异中,超过 98% 是非编码变异。而我们已有的工具往往仅专注于另外 2% 的基因组序列。
AlphaGenome
为了更准确、更快速且多模态、多维度地解码基因组,DeepMind 的研究人员开发了 AlphaGenome,它将多模态预测、长序列上下文和碱基对分辨率整合到一个框架中。
AlphaGenome 模型以长达 100 万个碱基对的长 DNA 序列作为输入,并预测数千种表征其调控活性的分子特性。它还可以通过比较突变序列与未突变序列的预测值来评估基因变异或突变的影响。
预测的特性包括基因在不同细胞类型和组织中的起始和终止位置、基因剪接的位置、产生的 RNA 数量,以及哪些 DNA 碱基可接近、彼此靠近或与某些蛋白质结合。
训练数据来自大型公共联盟,包括 ENCODE、GTEx、4D Nucleome 和 FANTOM5,这些联盟通过实验测量了这些特性,涵盖了数百种人类和小鼠细胞类型和组织中重要的基因调控模式。
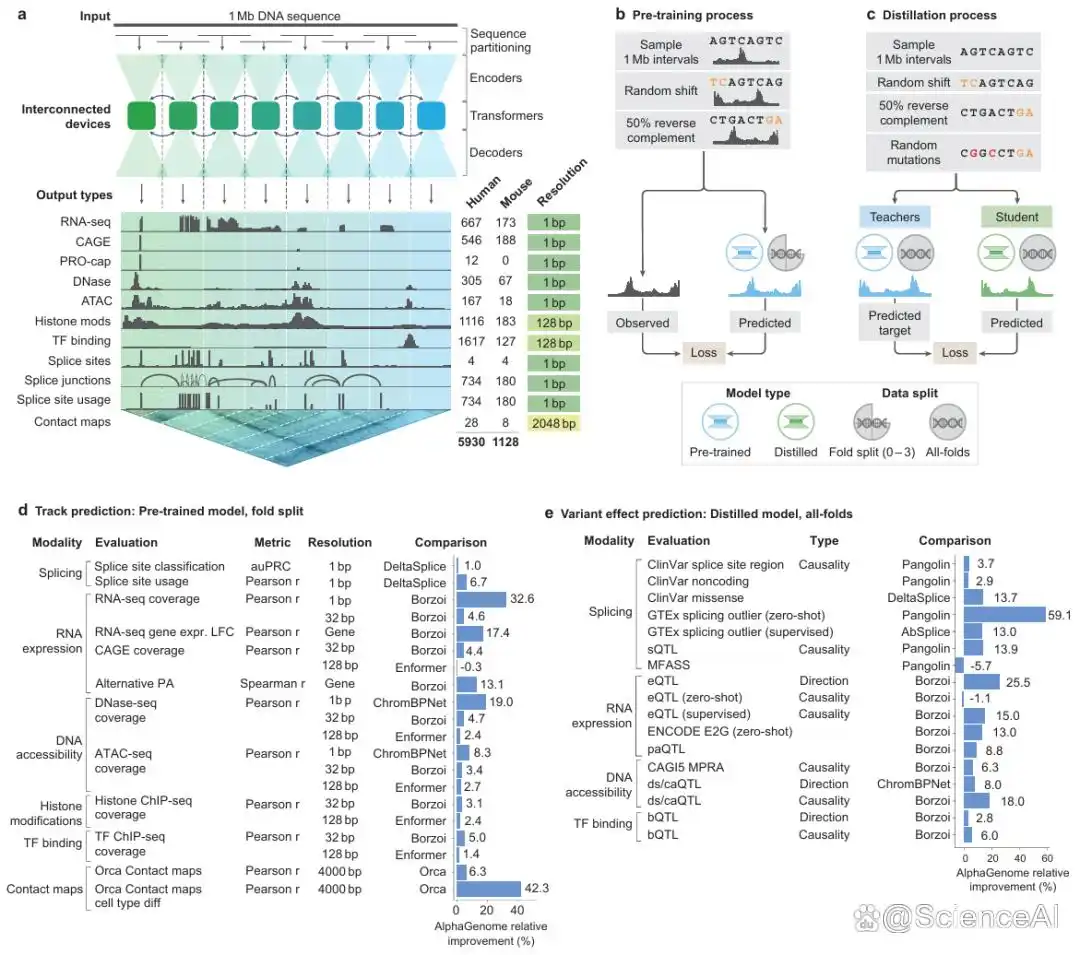
图示:AlphaGenome 模型架构、训练机制和综合评估性能。(来源:论文)
AlphaGenome 架构使用卷积层初步检测基因组序列中的短模式,使用 Transformer 在序列的所有位置传递信息,最后使用一系列层将检测到的模式转化为不同模态的预测。在训练期间,此计算分布在多个互连的张量处理单元 (TPU) 上,用于处理单个序列。
该模型基于 DeepMind 之前的基因组学模型 Enformer,并与 AlphaMissense 相辅相成,后者专门对蛋白质编码区内变异的影响进行分类。
强悍的性能
AlphaGenome 可以预测单个 DNA 字母的改变会如何影响基因表达,并改变其 RNA 和蛋白质产物。其他人工智能系统可以对蛋白质编码基因中约 2% 的基因组进行部分分析,而 AlphaGenome 是第一个对整个基因组进行同样分析的系统。
加州大学旧金山分校的 Hani Goodarzi 表示:「这是人工智能模型首次能够直接从 DNA 序列中准确预测 RNA(变体)的表达位置和方式。这不仅让我们能够了解基因是否表达,还能了解由此产生的 RNA 将如何被处理。」
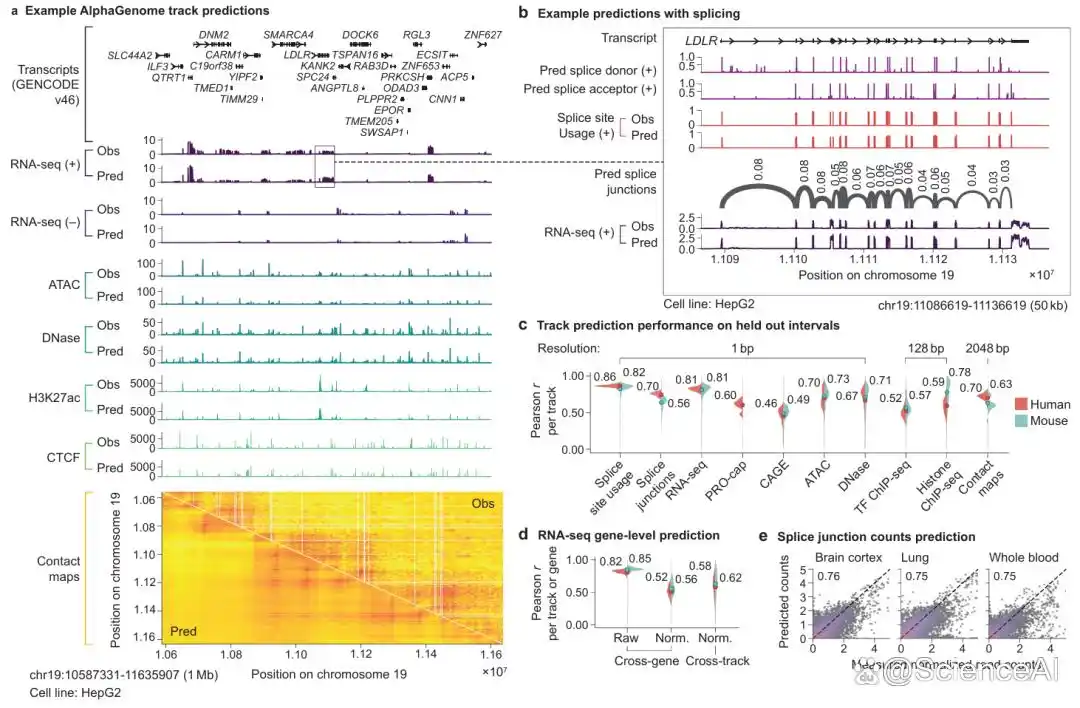
图示:AlphaGenome 轨迹预测和详细的性能评估。(来源:论文)
AlphaGenome 在人类和小鼠基因组上进行训练后,在 26 项变异效应预测评估中,有 24 项达到或超越了各自领域的最强外部模型;在 24 个基因组轨迹预测任务中,有 22 个取得了 SOTA 性能。它能够同时评估所有模态下的变异效应,从而准确地重现了 TAL1 致癌基因附近临床相关变异的机制。
伦敦大学学院的癌症分子生物学家 Marc Mansour 表示,当他的实验室将患者癌变组织细胞的基因组与未受影响细胞的基因组进行比较时,会发现数千个单独的碱基变异。「很难确定任何特定的变异是否会产生功能性后果。」Mansour 说道。很明显,TAL1 的案例证明 AlphaGenome 有潜力做到这点。
这种精确的定位功能「对我的研究至关重要。」Lareau 补充道,他的实验室分析基因变化对免疫功能的影响。「我不用测试数百个东西,而是可以专注于几个,这样就能找到正确的方向。」
研究人员表示 AlphaGenome 将在以下三个方面带来影响:
疾病理解:通过更准确地预测基因破坏,AlphaGenome 可以帮助研究人员更精准地查明疾病的潜在病因,并更好地解释与某些性状相关的变异的功能影响,从而可能发现新的治疗靶点。该模型尤其适用于研究可能产生巨大影响的罕见变异,例如导致罕见孟德尔遗传病的变异。
合成生物学:它的预测结果可用于指导具有特定调控功能的合成 DNA 的设计——例如,仅激活神经细胞中的基因,而不激活肌肉细胞中的基因。
基础研究:它可以通过协助绘制基因组的关键功能元件图谱并明确其作用,识别调控特定细胞类型功能的最重要 DNA 指令,从而加速生物学家对基因组的理解。
未来方向
尽管如此,AlphaGenome 仍然存在局限性。
与其他基于序列的模型一样,准确捕捉非常远的调控元件(例如距离超过 10 万个 DNA 碱基的调控元件)的影响仍然是一个持续的挑战。研究人员表示,未来研究的另一个重点是进一步提升该模型捕捉细胞和组织特异性模式的能力。
另外,该团队尚未设计或验证 AlphaGenome 用于个人基因组预测,这是 AI 模型面临的一个已知挑战。相反,他们目前更专注于表征其在个体基因变异上的表现。
虽然 AlphaGenome 可以预测分子结果,但它并不能全面展现基因变异如何导致复杂的性状或疾病。这些通常涉及更广泛的生物学过程,例如发育和环境因素,而这些内容已经超出了该模型的直接研究范围。这也反映了另一个方向,就是需要更多团队参与这项研究。
目前,AlphaGenome 已开放非商业使用,可以通过 AlphaGenome API 获取。
AlphaGenome:https://github.com/google-deepmind/alphagenome
最后还有个问题,这是否会让不法分子更容易设计生物武器呢?DeepMind 研究副总裁 Pushmeet Kohli 表示,公司已与外部生物安全专家分享了该模型。
「我们收到的反馈是,它非常安全,发布它的好处远远大于风险。」Kohli 补充说,DeepMind 希望继续扩展 AlphaGenome 的功能,例如提供更深入的洞察,帮助人们理解基因变异如何导致复杂的性状或疾病。
「我们今天所拥有的就像 AlphaFold1 诞生时一样——迈出了重要的第一步。」Kohli 说。





